找出网站所有死链接网站建设项目报告
最近 Chrome 117,CSS 又悄悄推出了一个新的的@规则,叫做@starting-style。从名称上来看,表示定义初始样式。那么,具体是做什么的?有什么用?一起了解一下吧
一、快速了解 @starting-style
通常做一个动画效果,你可能会考虑 transition 和 animation。
相对于animation,transition使用更简单,但是有一定条件,需要有状态的改变,例如手动添加一个class
div{transform: scale(0)
}
div.show{transform: scale(1)
}
示意如下
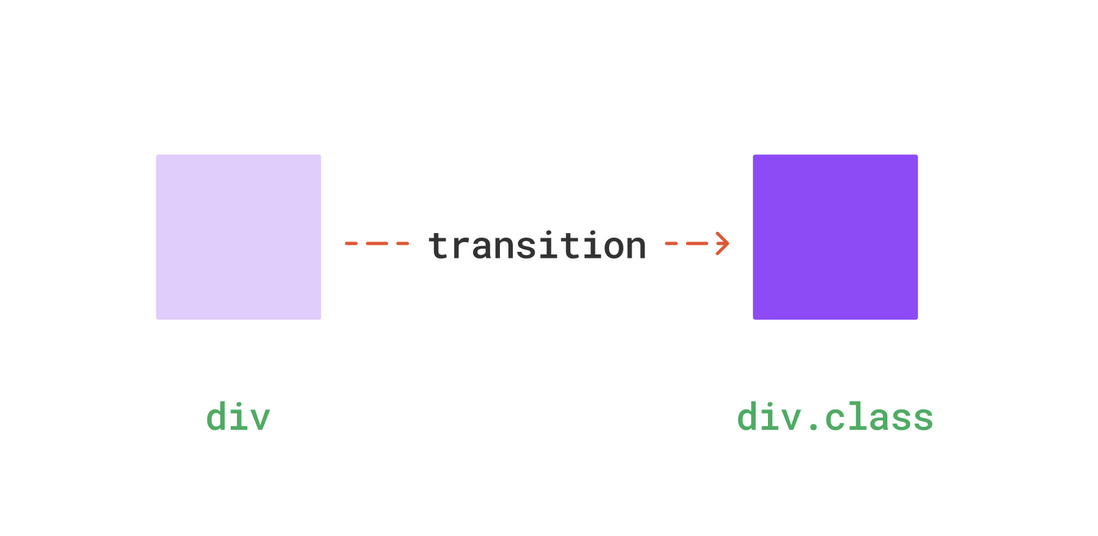
但是,如果这个show是一开始就存在的,比如
<div class="show"></div>
这样在页面打开的时候,肯定也是没有过渡效果的,因为没有状态的变化。
在以前,我们可以换成用animation的方式,这样即使是一开始存在的,也能有动画效果,因为animation是可以自动运行的。
不过到了现在,我们可以用transition的方式来实现了,将上面的例子改写一下
div{transform: scale(1);transition: 1s;
}
@starting-style {div{transform: scale(0);}
}
这里的@starting-style表示初始样式,相当于在渲染之前就有了一个初始状态,这样也就算有状态变化了
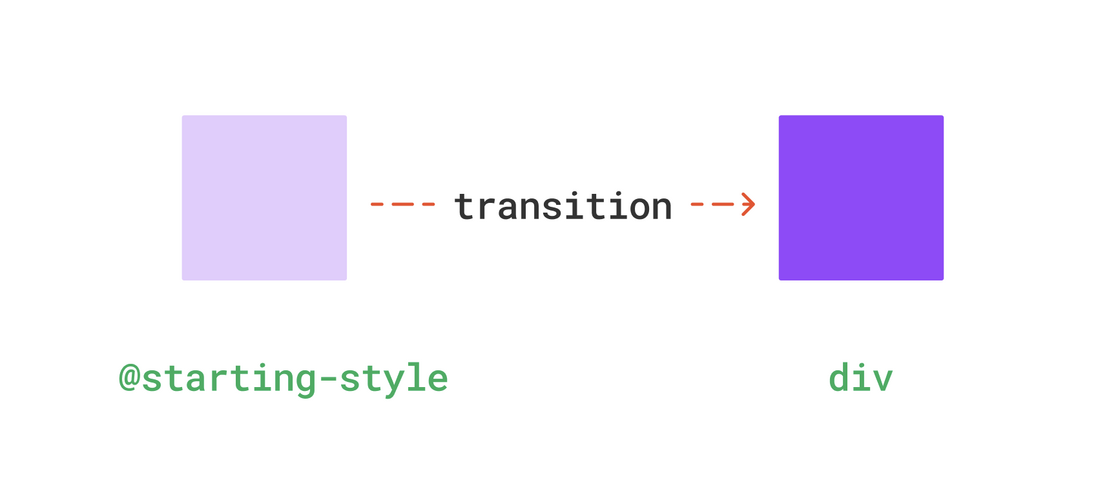
实际效果如下(每次刷新浏览器都有放大动画)

这样,即使不手动添加状态也能触发过渡动效了,这就是@starting-style。
二、元素添加时添加过渡
有时候,即使是手动添加class,也无法保证一定能触发过渡动效,比如新创建的元素
const div = document.createElement('div')
div.className = 'show' //过渡无效,直接就生效了
document.body.append(div)
这种情况下,transition就失效了,因为你在添加class的时候元素还未完全渲染。
要解决这个问题,之前也有几种方式
首先是定时器,添加一点点延时
settimeout(()=>{div.className = 'show'
},50)
还有一种方式,主动触发元素的渲染,强制重绘
div.clientWidth // 强制触发重绘 div.className = 'show'
另外,还可以用动画animation来代替,这样也能主动触发动画
.show{animation: ...
}
现在,使用@starting-style也能实现这样的效果
div.show{transition: 1s;
}
@starting-style {div{transform: scale(0);}
}
下面是一个元素出现过渡效果

你也可以访问以下链接查看实际效果(Chrome 117+)
- CSS @style-rule (codepen.io)')点击预览
这让我想起了之前做过一个message效果,实现原理是这样的,如果页面上还没有 message元素,就先创建,然后添加show类名,让这个元素出现,这里就是通过强制触发重绘实现的
function showMessage(txt){this.timer && clearTimeout(this.timer);var oDiv = document.getElementById('messageInfo');if(!oDiv){oDiv = document.createElement('div');oDiv.className = 'messageInfo';oDiv.id = 'messageInfo';document.body.appendChild(oDiv);}oDiv.innerHTML = '<span>'+txt+'</span>';div.clientWidth; // 强制触发重绘oDiv.classList.add('show');this.timer = setTimeout(function(){oDiv.classList.remove('show');},2000)
}
效果如下,第一次创建的时候也有过渡效果

有兴趣的可以回顾之前这篇4年前的文章:css3元素出现动画实例
三、让 display:none 也支持过渡
大家可能知道,当一个元素从display:none变成display:block时,是无法触发过渡效果的,即便有一些过渡属性
div{display: none;transition: 1s;transform: scale(0)
}
div.show{display: block;transform: scale(1)
}
像这种情况下没有过渡效果的,如下

不过,现在有了@starting-style,也能轻易实现过渡效果,不管你有没有display:none
/*仅需添加一个初始状态*/
@starting-style {div{transform: scale(0);}
}
效果如下

遗憾的是,从display:block变为display:none是无法触发过渡效果的
另外,原生组件很多的隐藏和显示都是直接通过display:none实现的,例如dialog,可以直接添加@starting-style规则来实现打开动画,而无需改变默认 display
dialog{transition: 1s;
}
@starting-style {dialog{transform: scale(0);}
}
效果如下

你也可以访问以下链接查看实际效果(Chrome 117+)
- CSS @style-rule display (codepen.io)')
四、总结一下
一个可以改变元素初始状态的新特性,你学到了吗?
- transition 需要有状态的改变才能触发过渡效果
- animation 无需状态改变,因为可以自动运行
- @starting-style 可以改变元素的初始状态,让元素在初次渲染时也有过渡效果
- @starting-style 可以在元素添加时直接添加过渡效果
- @starting-style 可以让 display:none 也支持过渡
不过像这样的 CSS 特性注定是冷门属性,主要是可替代性太强了,而且不知道什么时候才可以正式投入使用,现在就当提前了解吧。最后,如果觉得还不错,对你有帮助的话,欢迎点赞、收藏、转发 ❤❤❤
